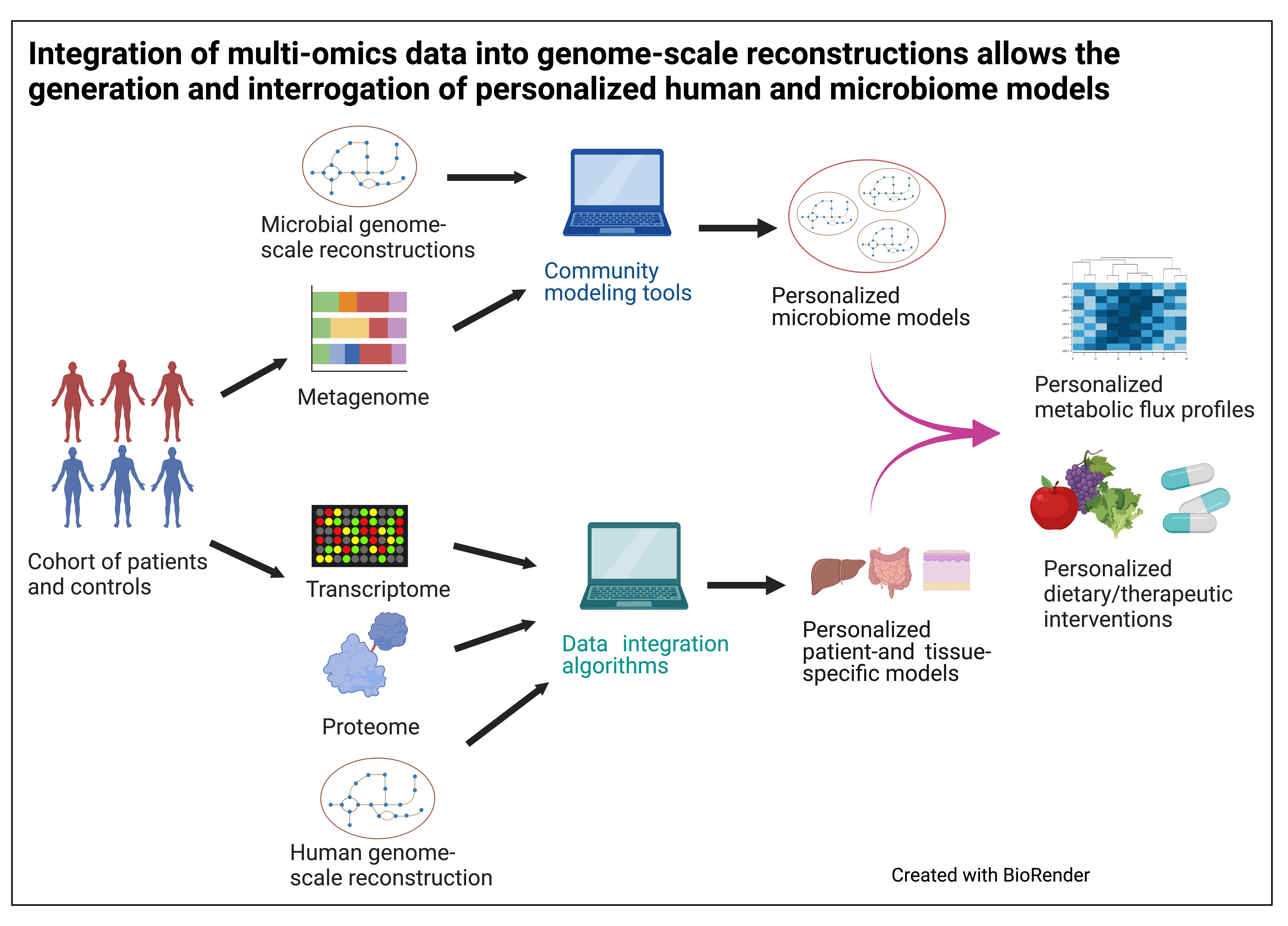
Decription du coursLe développement de méthodes omiques à haut débit telles que la génomique, la transcriptomique, la protéomique et la métagénomique a conduit à une avalanche de données disponibles. Cela a motivé le domaine de la biologie des systèmes, qui vise à caractériser de manière exhaustive les composants des systèmes biologiques tels que les cellules, les organismes ou les populations afin de comprendre leur fonction et d'interpréter les données omiques dans leur contexte biologique. Une approche puissante de la biologie des systèmes est la reconstruction et l'analyse basées sur les contraintes (COBRA). La méthode COBRA repose sur des reconstructions à l'échelle du génome affinées manuellement, qui peuvent être converties en modèles mathématiques et interrogées au travers de simulations. L'intégration de données transcriptomiques, protéomiques ou métagénomiques dans les modèles à l'échelle du génome permet de générer des modèles personnalisés qui peuvent segmenter les échantillons en fonction des flux métaboliques calculés. La modélisation personnalisée pourrait permettre de mieux comprendre les mécanismes d’une maladie et a des applications précieuses en médecine de précision, comme la conception de traitements ciblés. Dans ces cours, les méthodes de pointe visant l'intégration des données omiques dans les modèles à l'échelle du génome seront enseignées. Un des cours sera fondé sur l'utilisation des données transcriptomiques et protéomiques dans la construction de modèles contextuels et spécifiques au tissu donné. Les applications de ces modèles dans l'analyse d’états pathologiques ainsi que dans la médecine de précision seront examinées. Des tutoriels pratiques permettront aux participants d'appliquer des algorithmes pour la génération de modèles contextuels à leurs données. Un autre cours abordera l’utilisation de données métagénomiques afin de construire des modèles communautaires spécifiques au microbiome. Les applications récentes de la modélisation des communautés microbiennes pour élucider les interactions entre l'hôte et le microbiome dans les maladies seront passées en revue. Des tutoriels permettront aux participants d'acquérir une expérience pratique de l'intégration des données métagénomiques dans les modèles de communautés microbiennes et de prédire le métabolisme au niveau de la communauté. La formation se terminera par une table ronde autour de laquelle les participants pourront discuter des différentes stratégies et défis de leur recherche. Objectifs d’apprentissage spécifiques : - Concepts de base de la biologie des systèmes et de la modélisation basée sur les contraintes - Génération de reconstructions de haute qualité à l'échelle du génome - Méthodes de génération de modèles contextuels à partir de données transcriptomiques et protéomiques - Méthodes de modélisation métabolique des communautés microbiennes - Modélisation des interactions entre l'homme et le microbiome - Applications en médecine de précision
|